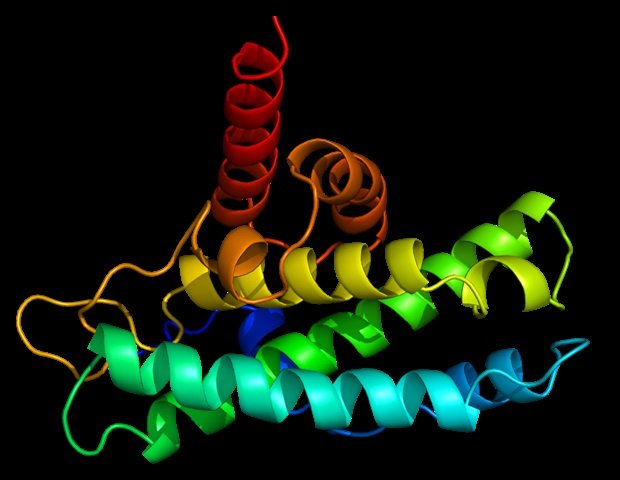
Des chimistes du MIT ont découvert la structure d’une protéine capable de pomper des molécules toxiques hors des cellules bactériennes. Des protéines semblables à celle-ci, que l’on retrouve dans E. colisont censés aider les bactéries à devenir résistantes à plusieurs antibiotiques.
Grâce à la spectroscopie par résonance magnétique nucléaire (RMN), les chercheurs ont pu déterminer comment la structure de cette protéine change lorsqu’une molécule de type médicament se déplace à travers elle. La connaissance de cette structure détaillée pourrait permettre de concevoir des médicaments susceptibles de bloquer ces protéines de transport et d’aider à resensibiliser les bactéries résistantes aux antibiotiques existants, explique Mei Hong, professeur de chimie au MIT.
Connaissant la structure de la poche de liaison aux médicaments de cette protéine, on pourrait essayer de concevoir des concurrents à ces substrats, de façon à bloquer le site de liaison et empêcher la protéine d’éliminer les antibiotiques de la cellule.”
Mei Hong, professeur de chimie au MIT, auteur principal de l’article.
Alexander Shcherbakov, étudiant diplômé du MIT, est l’auteur principal de l’étude, qui paraît aujourd’hui dans la revue Nature Communications. L’équipe de recherche comprend également Aurelio Dregni, étudiant diplômé du MIT, et deux chercheurs de l’université du Wisconsin à Madison : Peyton Spreacker, étudiant diplômé, et Katherine Henzler-Wildman, professeur de biochimie.
Transporteurs de résistance aux médicaments
Le pompage de médicaments à travers leurs membranes cellulaires est l’une des nombreuses stratégies que les bactéries peuvent utiliser pour échapper aux antibiotiques. Depuis plusieurs années, le groupe de Henzler-Wildman à l’Université du Wisconsin étudie une protéine membranaire appelée EmrE, qui transporte potentiellement de multiples molécules toxiques différentes, notamment des herbicides et des composés antimicrobiens.
EmrE appartient à une famille de protéines appelée les petits transporteurs SMR (multidrug resistance). Bien qu’EmrE ne soit pas directement impliqué dans la résistance aux antibiotiques, d’autres membres de cette famille ont été trouvés dans des formes résistantes aux médicaments de l’organisme. Mycobacterium tuberculosis et Acinetobacter baumanii.
“Les transporteurs SMR ont une conservation de séquence élevée dans les régions clés de la protéine. EmrE est de loin le membre le plus étudié de la famille, en même temps pour les raisons suivantes in vitro vivoce qui en fait un système modèle idéal pour étudier la structure qui soutient l’activité du SMR”, explique Henzler-Wildman.
Depuis quelques années, le laboratoire d’Hong a mis au point une technique qui permet aux chercheurs d’utiliser la RMN pour mesurer les distances entre les sondes de fluor et les atomes d’hydrogène dans les protéines. Il est ainsi possible de déterminer la structure d’une protéine lorsqu’elle se lie à une molécule contenant du fluor.
Après que Hong a présenté sa nouvelle technique lors d’une conférence, Henzler-Wildman a suggéré qu’ils fassent équipe pour étudier EmrE. Son laboratoire a passé de nombreuses années à étudier comment EmrE transporte une molécule de type médicament, ou ligand, à travers la membrane phospholipidique. Ce ligand, connu sous le nom de F4-TPP+est une molécule tétraédrique à laquelle sont attachés quatre atomes de fluor, un à chaque coin.
En utilisant ce ligand avec la nouvelle technique RMN de Hong, les chercheurs ont entrepris de déterminer une structure à résolution atomique de l’EmrE. On savait déjà que chaque molécule d’EmrE contient quatre hélices transmembranaires à peu près parallèles. Deux molécules EmrE s’assemblent en un dimère, de sorte que huit hélices transmembranaires forment des parois internes qui interagissent avec le ligand lors de son déplacement dans le canal. Des études antérieures ont révélé la topologie globale des hélices, mais pas des chaînes latérales de la protéine qui s’étendent à l’intérieur du canal, qui sont comme des bras qui saisissent le ligand et aident à le guider dans le canal.
EmrE transporte des molécules toxiques de l’intérieur d’une cellule bactérienne, dont le pH est neutre, vers l’extérieur, qui est acide. Ce changement de pH à travers la membrane affecte la structure d’EmrE. Dans un article publié en 2021, Hong et Henzler-Wildman ont découvert la structure de la protéine lorsqu’elle se lie à F4-TPP+ dans un environnement acide. Dans le nouveau Nature Communications , ils ont analysé la structure à un pH neutre, ce qui leur a permis de déterminer comment la structure de la protéine change en fonction du pH.
Une structure complète
À pH neutre, les chercheurs ont constaté dans cette étude que les quatre hélices qui composent le canal sont relativement parallèles les unes aux autres, créant une ouverture dans laquelle le ligand peut facilement entrer. Lorsque le pH baisse, en se déplaçant vers l’extérieur de la membrane, les hélices commencent à s’incliner de sorte que le canal est plus ouvert vers l’extérieur de la cellule. Cela permet de pousser le ligand hors du canal. Dans le même temps, plusieurs anneaux présents dans les chaînes latérales des protéines changent d’orientation d’une manière qui contribue également à guider le ligand hors du canal.
L’extrémité acide du canal est aussi plus accueillante pour les protons, qui entrent dans le canal et l’aident à s’ouvrir davantage, permettant au ligand de sortir plus simplement.
“Cet article complète vraiment l’histoire”, dit Hong. “Une structure ne devient pas suffisante. Il en faut deux, pour comprendre comment un transporteur peut réellement s’ouvrir des deux côtés de la membrane, car il est censé pomper le ligand ou le composé antibiotique de l’intérieur de la bactérie vers l’extérieur.”
On pense que le canal EmrE transporte de nombreux composés toxiques différents. Hong et ses collègues prévoient donc d’étudier comment d’autres molécules voyagent à travers ce canal.
La recherche a été financée par les National Institutes of Health et le MIT School of Science Camplan Fund.
Source :
Institut de technologie du Massachusetts
Référence du journal :
Shcherbakov, A.A., et al. (2022) La structure à pH élevé de EmrE révèle le mécanisme de transport de substrat couplé au proton. Nature Communications. doi.org/10.1038/s41467-022-28556-6.




